揭秘TRAF与TAK基因的高效克隆技术
TRAF基因和TAK基因的克隆方法

TRAF(TNF Receptor Associated Factor)基因和TAK(TGF-β-activated Kinase)基因在细胞信号传导过程中起着重要作用。为了全面理解这两种基因的克隆方法,我们需要从引物设计、基因扩增、载体连接、阳性克隆筛选以及测序分析等多个环节进行详细阐述。

一、实验材料与试剂
1. 模板DNA:含有TRAF和TAK基因的鱼类组织或细胞。

2. 引物:根据GenBank中已提交的鱼类TRAF6和TAKI cDNA序列的保守区设计的兼并引物。
3. PCR试剂:高保真DNA聚合酶、dNTPs、PCR buffer、MgCl₂等。

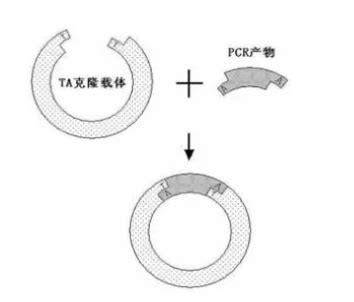
4. 载体:PMD18-T载体(TaKaRa, Japan)。
5. 感受态细胞:trans5a大肠杆菌。
6. 电泳试剂:1%琼脂糖凝胶、电泳缓冲液(如TAE或TBE)。
7. 测序服务:提供高质量的测序服务。
二、实验步骤
1. 引物设计与合成
首先,从GenBank中检索鱼类TRAF6和TAKI基因的cDNA序列,分析其保守区,设计兼并引物(TRAF6-F/TRAF6-R和TAKI-F/TAKI-R)。引物设计时需注意引物的长度、GC含量、退火温度等参数,以确保PCR扩增的高效性和特异性。
2. PCR扩增基因核心片段
以模板DNA为模板,使用设计的兼并引物进行PCR扩增。PCR反应体系通常包括模板DNA、引物、高保真DNA聚合酶、dNTPs、PCR buffer和MgCl₂等。在适当的退火温度和延伸时间下,进行PCR扩增,以获得TRAF6和TAKI基因的核心片段。
PCR产物需经过1%琼脂糖凝胶电泳检测,确认扩增产物的大小和特异性。将扩增产物进行纯化,以便后续步骤的连接。
3. 载体连接与转化
将纯化后的PCR产物与PMD18-T载体进行连接。连接反应体系通常包括PCR产物、PMD18-T载体、T4 DNA连接酶和连接buffer。在适当的温度下,进行连接反应,将PCR产物插入到PMD18-T载体中。
连接产物转化到trans5a感受态细胞中。转化过程包括冰浴、热激和复苏等步骤。转化后的细胞涂布在含有抗生素的平板上,进行培养。
4. 阳性克隆筛选与测序
从平板上挑取单菌落,进行PCR检测,确认是否含有插入片段。PCR检测通常使用载体上的通用引物和目的基因的一个特异性引物。将阳性克隆进行测序,以获取TRAF6和TAKI基因的核心片段序列。
测序结果通过BLAST比对分析,确认扩增的片段是否为TRAF6和TAKI基因的核心片段。
5. RACE扩增基因全长
根据获得的TRAF6和TAKI基因的核心片段,设计RACE特异性引物。利用巢式PCR扩增TRAF6和TAKI基因的5'和3'末端。RACE扩增包括第一轮PCR和第二轮PCR,使用不同的引物和退火温度。
PCR产物同样需要经过1%琼脂糖凝胶电泳检测,确认扩增产物的大小和特异性。将扩增产物连接到载体上,转化到trans5a感受态细胞中,筛选阳性克隆进行测序。
测序结果通过BLAST比对分析,确认扩增的片段是否为TRAF6和TAKI基因的全长序列。使用Seqman软件拼接得到的序列,以获得完整的TRAF6和TAKI基因序列。
6. 序列比对与系统进化分析
利用EditSeq软件预测TRAF6和TAKI基因的开放阅读框(ORF)及可能编码的氨基酸序列。将预测的氨基酸序列与其他物种的TRAF6和TAKI基因的氨基酸序列进行比对,分析序列的保守性和差异性。
在NCBI数据库中搜寻其他物种的TRAF6和TAKI的氨基酸序列,利用MEGAS.O软件,以Neighbor jining法构建系统进化树。系统进化树可以展示不同物种TRAF6和TAKI基因的进化关系和亲缘关系。
三、注意事项
1. 引物设计:引物设计时需注意引物的特异性、长度和退火温度等参数,以确保PCR扩增的高效性和特异性。
2. PCR扩增:PCR扩增过程中需注意模板DNA的质量、PCR反应体系的组成和扩增条件的优化,以获得高质量的扩增产物。
3. 载体连接:载体连接过程中需注意连接酶的选择、连接反应体系的组成和连接条件的优化,以确保连接的高效性和准确性。
4.
- 上一篇: 街舞的多样魅力:探索那些燃爆舞池的种类
- 下一篇: 圆通快递收费标准详解
-
 揭秘:杂交水稻与转基因,它们是一回事吗?资讯攻略11-02
揭秘:杂交水稻与转基因,它们是一回事吗?资讯攻略11-02 -
 生活中我们常遇到的10种转基因食品都有哪些?资讯攻略10-31
生活中我们常遇到的10种转基因食品都有哪些?资讯攻略10-31 -
 轻松掌握:鉴别转基因食品的实用指南!资讯攻略11-05
轻松掌握:鉴别转基因食品的实用指南!资讯攻略11-05 -
 解锁DBsystem龙之气息:核心系统深度攻略,揭秘提升基因突变好运的终极秘诀!资讯攻略10-26
解锁DBsystem龙之气息:核心系统深度攻略,揭秘提升基因突变好运的终极秘诀!资讯攻略10-26 -
 探索基因世界的奥秘:等位基因与非等位基因的区别资讯攻略11-07
探索基因世界的奥秘:等位基因与非等位基因的区别资讯攻略11-07 -
 有哪些已经灭绝的动物被成功复活了?资讯攻略11-10
有哪些已经灭绝的动物被成功复活了?资讯攻略11-10












